Adaptation des bactéries à leur milieu / environnement
Interstell’Art
Fun fact : Si toutes les bactéries de la Terre étaient mises en file indienne, elles s’étaleraient sur une distance de 10 milliards d’année lumières, ce qui correspond à deux allers-retours entre la terre et le plus gros trou noir de l’univers (galaxie centrale d’Abell 2199 situé à 590 millions d’années lumières de la voir lactée).
UT3 : estelle.crozat-brendon@univ-tlse3.fr
Nathalie Campo, Chercheure CNRS : nathalie.campo@univ-tlse3.fr
Isabelle Mortier, Ingénieure de recherche CNRS :
isabelle.mortier@univ-tlse3.fr
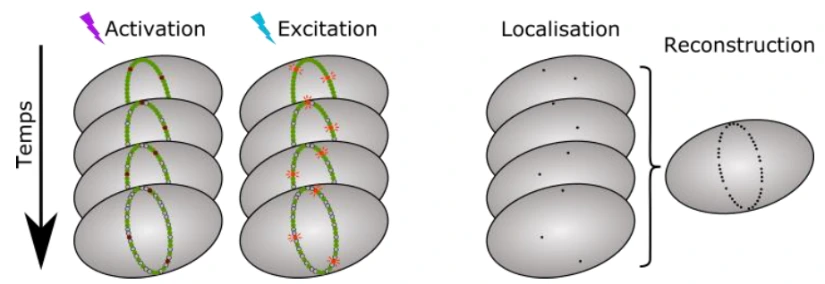
Nano-structure en trois dimensions de l’anneau de division « FtsZ » de Streptococcus pneumoniae. Cette image est une reconstruction obtenue par la technique de microscopie à super-résolution PALM (PhotoActived Localization Microscopy). Le PALM fait partie des techniques de microscopie basées sur la localisation de molécules uniques (Single Molecule Localization Microscopy) dont le principe consiste à exciter des molécules (les fluorophores) individuellement. La localisation de chaque molécule est détectée avec précision grâce à l’utilisation de logiciels d’analyse d’image adaptés. L’image finale correspond à la superposition de toutes les molécules uniques détectées. Échelle : 500 nm
Crédits photos : ©Isabelle Mortier-Barrière et ©Sylvain Cantaloube

Escherichia coli
Famille : Enterobacteriaceae
Forme : bâtonnet
Type : Gram négatif
Découverte : en 1885, par Theodor Escherich
Adresse : tube digestif
Spécificité : bactérie commensale dont la plupart des souches sont inoffensives, celles produisant des shigatoxines sont pathogènes.
Pathologies : gastroentérite, infection urinaire, méningite ou sepsis
Explications
Les bactéries sont de fabuleuses petites machines qui ont un pouvoir d’adaptation à leur environnement phénoménal. On en retrouve ainsi partout, depuis le fond des lacs subglaciaires de l’Antarctique, sur les racines des plantes, et en multitudes dans nos intestins.
Ces modes de vie très différents ont pourtant en commun des mécanismes de prolifération et de conservation de l’information génétique très similaires. Cette conservation est orchestrée au cours du cycle cellulaire qui comprend les étapes de réplication du matériel génétique (ADN), de ségrégation (séparation) des chromosomes frères dans chacune des cellules filles et de division en 2 cellules filles.
Au LMGM-CBI, l’équipe Dynamique des Génomes : Stabilité, Transfer et Intégration dans le cycle cellulaire et l’équipe Compétence et Transformation du pneumocoque, étudient le cycle cellulaire dans deux modèles bactériens différents.
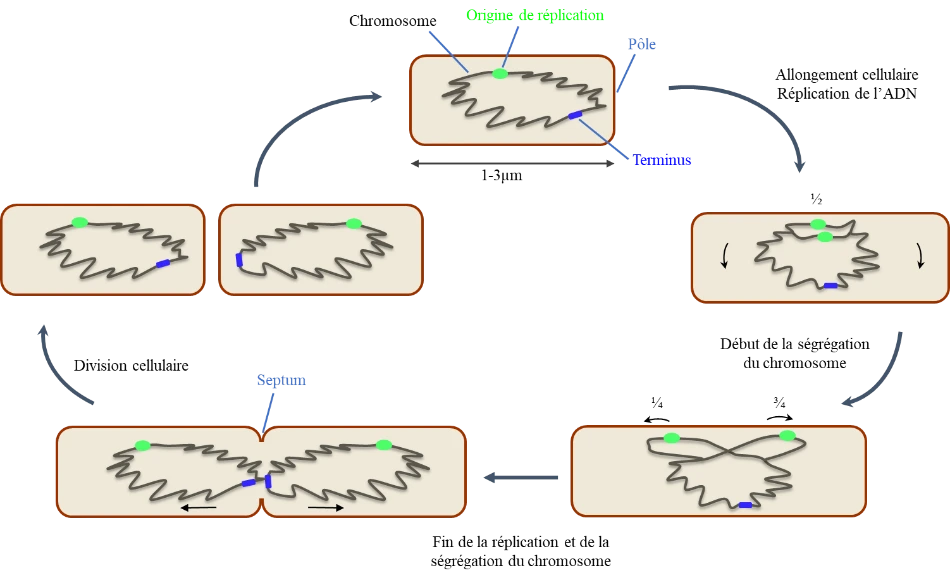
Le cycle cellulaire d’Escherichia coli
Les bactéries prolifèrent essentiellement par fission binaire, ce qui implique que la cellule, qui peut être de forme très variable, commence par s’allonger.
E. coli a une forme de bâtonnet, dont la longueur varie suivant le milieu de croissance et le stade du cycle cellulaire (voir figure du cycle cellulaire). Cette étape d’élongation (allongement) consiste tout d’abord à synthétiser de la membrane et du peptidoglycane (PG), macro-molécule qui protège et rigidifie la bactérie.
Au même moment, l’information génétique est dupliquée. Comme pour tous les êtres vivants, cette information se trouve dans une ou plusieurs molécules d’ADN, qui sont pour la majorité des bactéries sous une forme circulaire. Les bactéries contiennent un chromosome principal (la plus grande molécule d’ADN), et peuvent contenir d’autres chromosomes, ou des molécules d’ADN circulaires encore plus petites, appelées plasmides. Le chromosome est représenté sur la figure comme un cercle, mais il est important de réaliser que sa longueur est 1000 fois celle de la longueur de la cellule ! L’organisation de ce chromosome est donc primordiale pour le fonctionnement de la cellule.
Toutes ces molécules d’ADN doivent être copiées de manière fidèle avant toute division cellulaire, de manière à distribuer exactement la même information génétique à toutes les descendantes de la bactérie mère. Ce processus s’appelle la réplication. Elle est très fidèle (seulement 1 erreur pour 10 millions de bases incorporées !) et très rapide (environ 1000 bases/s). Ainsi, le chromosome d’E. coli, qui contient 4,6 millions de paires de bases, est copié en 40 min ! La réplication débute à un endroit précis du chromosome, appelé origine de réplication (en vert sur la figure), et s’étend de manière bidirectionelle, vers le terminus de réplication (en bleu sur la figure).
Cette organisation est également spatiale, puisque chaque partie du chromosome (appelée domaine), est localisée à un endroit bien spécifique de la cellule. Avant le début de la réplication, l’origine est située au centre de la cellule. Après sa réplication, les origines migrent vers les positions ¼ et ¾ de la cellule, qui correspondent aux centres des futures cellules filles. Le terminus, lui, se rapproche du centre de la cellule (appelé Septum) pendant la réplication, et reste à cette position après la division. Il se retrouve ainsi au pôle de la cellule fille.
Ce mouvement des chromosomes répliqués vers les deux cellules filles est appelé ségrégation. Il est en effet indispensable que chacune des copies de l’information génétique soit distribuée de manière égale dans les 2 cellules en formation. Seul le terminus reste au niveau du septum jusqu’à sa fermeture. Des signaux moléculaires indiquent en effet à la cellule que la réplication est terminée, la ségrégation est faite, et donc le septum peut se fermer, d’une manière analogue à un anneau qui se resserre. Cela aboutit à sa fermeture, et à la séparation des deux cellules filles.
Un nouveau cycle cellulaire peut alors commencer.
Un exemple de détection directe de transfert horizontal au microscope. La transformation de L’ADN marqué en rouge permet ici la production d’une structure verte dans les cellules.
Une des étapes du cycle cellulaire est la division. La division est un processus fondamental du cycle cellulaire qui en coordination avec la réplication et la ségrégation du chromosome, comprend différentes étapes finement régulées qui aboutissent à la formation de deux cellules filles.
L’équipe Compétence et Transformation du pneumocoque, s’intéresse particulièrement à ce processus chez Streptococcus pneumoniae.
La division du pneumocoque
Comme pour Escherichia coli, la division est un processus fondamental du cycle cellulaire du pneumocoque. Elle requiert l’assemblage d’un complexe multiprotéique appelé le « divisome ». La localisation de ce complexe au site de division de la cellule mère est critique pour assurer la formation du septum de division et la constriction de la membranes cellulaire.
Détermination du site de division :
La protéine FtsZ (« Filamentous Temperature Sensitive Z »), présente chez presque toutes les bactéries, est une des protéines clé du processus de division. Les avancées technologiques réalisées ces dernières années ont permis de mieux comprendre son rôle et son fonctionnement, notamment grâce aux techniques de microscopie de super-résolution.
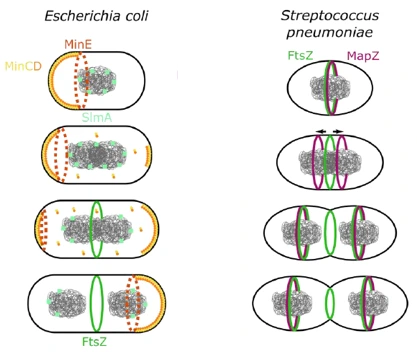
Chez les bacilles, la localisation au centre de la bactérie est guidée principalement par un processus de sélection négative. Le système Min, par exemple, empêche la formation du site de division près des pôles cellulaires ce qui empêche l’anneau Z de se former ailleurs qu’au centre de la cellule.
Régulation du positionnement de l’anneau Z
Régulation négative chez E. coli : MinC (cercle jaune) est recrutée par MinD (cercle orange clair) à la membrane et oscille entre les pôles cellulaires sous le contrôle de MinE (pointillés orange foncé).
Régulation positive chez S. pneumoniae : la protéine MapZ se localise aux futurs sites de division afin de recruter la protéine FtsZ et former l’anneau Z.
Certaines espèces ont mis en place d’autres dispositifs pour une régulation positive du positionnement de leur site de division comme c’est le cas chez le pneumocoque. Ce mécanisme repose sur la protéine MapZ (« Midcell Anchored protein Z ») dont la présence est nécessaire au bon positionnement de l’anneau Z. Cette protéine membranaire interagit avec le peptidoglycane et s’assemble sous forme d’anneau au centre de la cellule avant n’importe quelle autre protéine de la division. La localisation de MapZ permet alors le recrutement de FtsZ au centre de la cellule. Suite à l’assemblage de l’anneau FtsZ, l’anneau MapZ se sépare en deux anneaux qui vont migrer jusqu’au futur site de division des cellules filles, poussés par la synthèse du peptidoglycane périphérique (élongation).
L’anneau de division FtsZ.
FtsZ est un homologue bactérien de la tubuline eucaryote et elle polymérise (s’assemble) sous forme d’anneau au niveau du futur site de division. La formation de cette structure permet de recruter d’autres protéines pour former le divisome complet. Parmi ces protéines, on retrouve notamment celles qui sont impliquées dans la synthèse du peptidoglycane (PG), constituant majoritaire de la paroi. La synthèse du PG permet l’allongement de la cellule puis sa division.
Structure de l’anneau FtsZ chez S.pneumoniae
A gauche, anneau FtsZ-GFP observé en épi-fluorescence au cours du cycle cellulaire chez S. pneumoniae. L’échelle représente 1 μm. A droite, modèle actuel de l’anneau Z en trois dimensions (échelle non respectée). L’anneau se compose de différents filaments situés près de la membrane interne (IM).
La microscopie PALM permet d’obtenir une résolution théorique d’environ 20 nm, soit un gain d’un facteur dix par rapport à la microscopie à fluorescence classique.
C’est avec cette technique de pointe que l’anneau FtsZ présenté sur le panneau a été réalisé.
Structure de l’anneau Z.
L’amélioration des techniques de microscopie au cours des 25 dernières années, ont permis d’affiner et préciser la structure de l’anneau Z. Cet anneau, longtemps imaginé comme une forme lisse et continue formée par un filament unique et ininterrompu, qui se positionnerait en spirale le long de la membrane interne de la cellule, apparait comme un ensemble de courts filaments de FtsZ interagissant entre eux. Plus récemment encore, le développement de techniques de super-résolution a permis de dévoiler que l’anneau Z constitue une structure granuleuse et discontinue.
Ancrage à la membrane : La formation de l’anneau FtsZ requiert la présence d’autres protéines du divisome parmi lesquelles FtsA. FtsA est un homologue de l’actine qui colocalise avec FtsZ au septum en formant également un anneau. Cette protéine essentielle à la survie des bactéries se lie à la membrane et par son interaction avec la protéine FtsZ, permet l’ancrage de cette dernière à la membrane. FtsA serait nécessaire à la formation et la stabilisation de l’anneau Z. Chez S. pneumoniae, l’association de ces deux protéines permettrait la coordination des synthèses périphérique et septale du PG.
Fonction de l’anneau Z : Le rôle premier identifié pour l’anneau Z est l’assemblage du divisome. FtsZ est une des premières protéines à se positionner au niveau du site de division avant de recruter les autres protéines nécessaires au bon déroulement de ce processus. FtsZ servirait d’échafaudage pour l’assemblage et la construction d’un appareil de division complet et mature. Le rôle de FtsZ serait donc plutôt de diriger correctement l’assemblage de la machinerie de synthèse de PG septale afin de gérer la formation des pôles cellulaires. Un autre rôle majeur de FtsZ serait de générer la force nécessaire à la constriction de la membrane cytoplasmique.
Visualisation de l’anneau Z par microscopie optique de fluorescence :
La microbiologie est notamment née grâce à l’utilisation des outils de microscopie. Antoni Van Leeuwenhoek a été le premier à observer et décrire, via un microscope de son invention, différents micro-organismes qu’il nomma alors « animalcules ». Par la suite, la microscopie est devenue un outil très utilisé, notamment en biologie car elle permet l’obtention de nombreuses informations invisibles à l’œil nu.
Techniques de microscopie de super-résolution :
La microscopie PALM (PhotoActivated Localization Microscopy) fait partie de la catégorie des techniques de microscopie basées sur la localisation de molécules uniques.
Contrairement à la fluorescence où tous les fluorophores présents au sein de l’échantillon émettent un signal fluorescent de façon simultanée, le principe des techniques de localisation de molécules uniques consiste à exciter chaque fluorophore individuellement. Aucun signal ne se chevauche et les molécules séparées de moins de 200 nm peuvent être différenciées. Les coordonnées spatiales de chaque fluorophore sont enregistrées de manière précise. L’excitation individuelle des fluorophores est réalisée par l’activation/excitation aléatoire d’une sous population des molécules présentes dans l’échantillon. Pour obtenir une image super-résolue, il faut donc procéder à des cycles successifs d’activation/excitation des fluorophores jusqu’à ce que la structure étudiée soit suffisamment échantillonnée ou qu’il n’y ait plus de molécule excitable dans l’échantillon. L’image finale correspond à une superposition de toutes les molécules uniques détectées au cours des cycles d’acquisition.