Les bactéries et la pathogénicité
L’effet ARN
Fun fact : La composition de ton microbiote est aussi unique que ton empreinte digitale.
Personne contact : Peter Redder, professeur de l’Université Toulouse III : peter.redder@univ-tlse3.fr
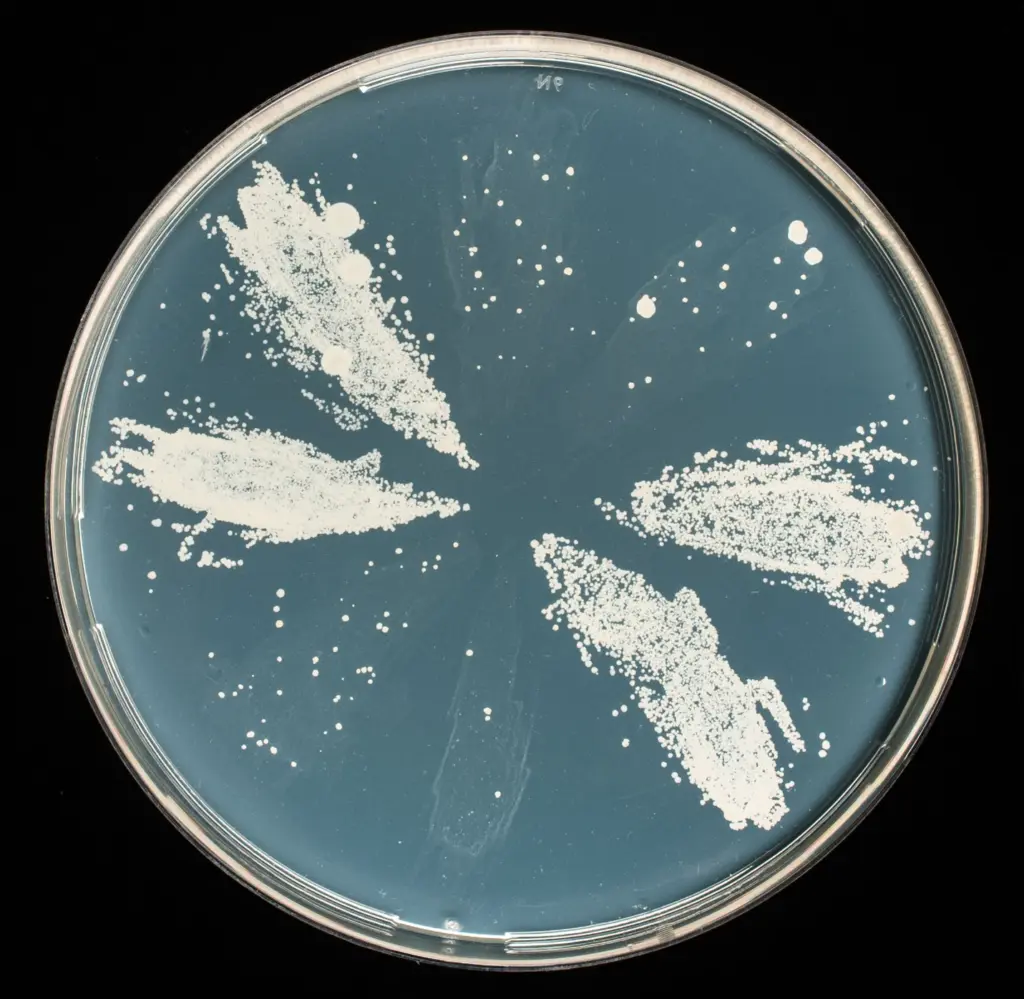
Expression de toxines chez Mycobacterium smegmatis
Expression de différentes toxines de type II de Mycobacterium tuberculosis clonées dans le plasmide pGMC (non réplicatif) et introduit dans le génome de Mycobacterium smegmatis par recombinaison homologue au niveau du gène du tRNA glyV.
Le plasmide pGMC a un promoteur inductible à l’anhydrotétracycline (ATc) et contient un gène de résistance à la streptomycine. Il s’intègre dans le chromosome de M. smegmatis par recombinaison homologue entre le site attP situé sur le plasmide et le site attB du chromosome par l’intermédiaire de la recombinase spécifique de site exprimée à partir du plasmide.
Les différents plasmides contenant les différentes toxines ont été introduits par électroporation dans des cellules compétentes de M. smegmatis.
Après transformation, les bactéries sont étalées sur des boites contenant du milieu LB gélosé et de la streptomycine à 25µg/mL. Les colonies apparaissent au bout de 3 jours à 37°C.
Crédits photos : @Moïse Mansour et @David Villa
Mycobacterium smegmatis
Staphylococcus aureus
Mycobacterium tuberculosis
Explications
Pourquoi étudier l’ARN ?
Dans les cellules, l’ADN est transcrit (lu) pour produire un ARN messager qui va ensuite être traduit en protéine. Ce processus est généré pour la production de chaque protéine dans toutes les cellules.
Chez les bactéries, la concentration cellulaire d’un ARN messager, et donc potentiellement le niveau d’expression de la protéine codée pas celui-ci, est déterminé par deux facteurs principaux : la transcription (lecture) et la stabilité (durée de vie) des ARN.
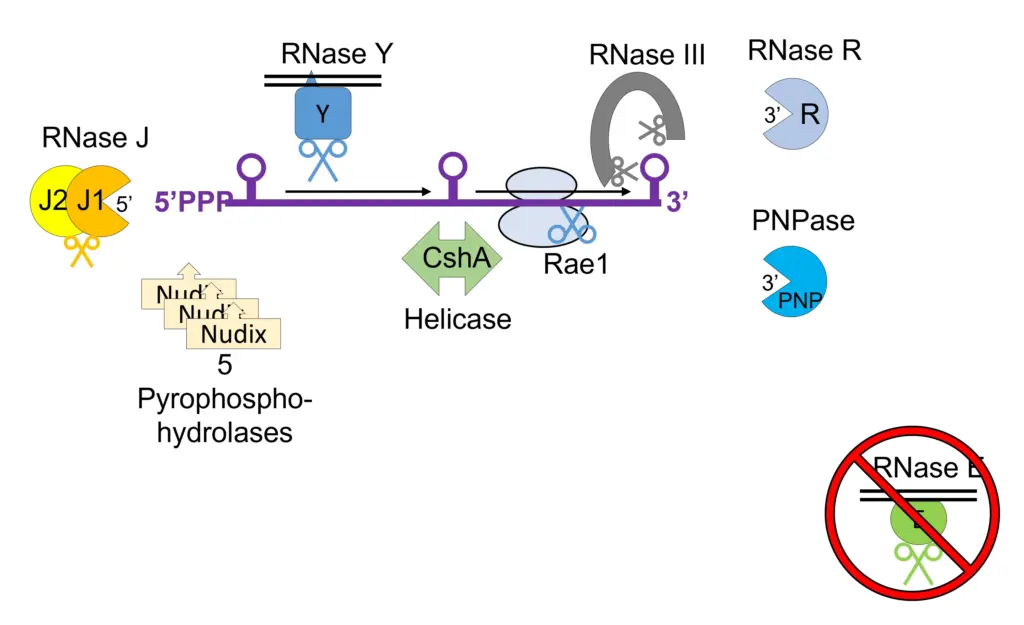
La stabilité des ARN dépend d’enzymes impliquées dans leur dégradation. Elles sont de deux types :
– les endo-ribonucléases (petits ciseaux sur le schéma) qui coupent l’ARN en morceaux plus petits
– les exo-ribonucléases (les « PacMan (bouches) » sur le schéma) qui dégradent l’ARN en enlevant les nucléotides un après l’autre à partir des extrémités.
Certaines enzymes ont les deux activités.
Pourquoi étudier l’ARN ?
Dans les cellules, l’ADN est transcrit (lu) pour produire un ARN messager qui va ensuite être traduit en protéine. Ce processus est généré pour la production de chaque protéine dans toutes les cellules.
Chez les bactéries, la concentration cellulaire d’un ARN messager, et donc potentiellement le niveau d’expression de la protéine codée pas celui-ci, est déterminé par deux facteurs principaux : la transcription (lecture) et la stabilité (durée de vie) des ARN.
La stabilité des ARN dépend d’enzymes impliquées dans leur dégradation. Elles sont de deux types :
– les endo-ribonucléases (petits ciseaux sur le schéma) qui coupent l’ARN en morceaux plus petits
– les exo-ribonucléases (les « PacMan (bouches) » sur le schéma) qui dégradent l’ARN en enlevant les nucléotides un après l’autre à partir des extrémités.
Certaines enzymes ont les deux activités.
Staphylococcus aureus très connu sous le nom « Staphylocoque doré ». Nous nous intéressons à cette bactérie pathogène chez qui la modification de la régulation de certains gènes entraine un fort impact sur la santé humaine. Staphylococcus aureus fait partie de la flore bactérienne normale d’environ 30% de population humaine. Mais, dans les conditions particulières, cette bactérie peut devenir un dangereux pathogène capable de causer une gamme de maladies très diverses : pneumonie, infections des os, infections des valves du cœur, mais aussi des infections superficielles comme l’impétigo ou les furoncles. Parce que S. aureus est partout dans notre société, il pose de graves problèmes dans le milieu hospitalier où Staphylococcus aureus est la première cause d’infections secondaires ou nosocomiales. En plus de son très haut risque d’infection, Staphylococcus aureus présente des problèmes importants de résistances aux antibiotiques. En effet, de nombreux variants (souches) ont été identifiés présentant des résistances contre un ou plusieurs antibiotiques, et certains d’entre eux avec des résistances contre tous les antibiotiques existants (multirésistants).
Pourquoi étudier l’ARN chez S. aureus ?
Afin de réguler de nombreux processus cellulaires comme le passage d’agent commensal (non pathogène et présent dans les microbiotes humains) à pathogène agressif, S. aureus nécessite une régulation génique précise qui ne peut être atteinte que si la demi-vie (disparition de 50%) des ARN est maintenue à une durée courte, généralement comprise entre 1 et 5 minutes. Chez S. aureus, l’une des enzymes clés de la dégradation des ARN est la RNase Y, une endoribonucléase associée à la membrane. Il a été montré que chez la souris, la suppression de la RNase Y entraine une détérioration de la virulence : S. aureus n’est plus aussi dangereux !
En conclusion, comprendre comment les ARN sont dégradés, va permettre de chercher de nouveaux modes de lutte contre S. aureus.